Neuerungen des diagnostischen Labors
Januar 2025
Erregernachweis bei Blutstrominfektionen mittels Sequenzierung von zellfreier DNA (Noscendo DISQVER®)
Neueinführung Testverfahren
Erregernachweis bei Blutstrominfektionen mittels Sequenzierung von zellfreier DNA (Noscendo DISQVER®)
DISQVER® ist ein Next-Generation-Sequencing-basierter, CE-zertifizierter diagnostischer Test zur Detektion von Blutstrominfektionen mittels zellfreier DNA aus Vollblut. Das Verfahren bietet eine hypothesenfreie Identifikation von Bakterien, Pilzen, DNA-Viren und Parasiten aus einer einzelnen Blutprobe. Dabei wird aus der Blutprobe die zellfreie DNA sequenziert und mit einer Datenbank abgeglichen, die aktuell ca. 16.000 Spezies umfasst. Da es sich um ein kulturfreies Nachweisverfahren handelt, können auch Erreger nachgewiesen werden, die in Standardkulturverfahren schwer anzuzüchten sind. Die Bereitstellung der CE-zertifizierten Analysesoftware und Datenbank erfolgt durch einen externen Dienstleister (NOSCENDO).
Seit April 2024 ist die Abteilung Medizinische Mikrobiologie für die Probenverarbeitung und Sequenzierung von zell-freier DNA zertifiziert. Durch die lokale Prozessierung der Proben können wir eine schnellere Verarbeitung der Proben ermöglichen.
Die Leistung wird im Rahmen der innerbetrieblichen Leistungsverrechnung mit der einsendenden Abteilung abgerechnet.
Für die Indikationsstellung sollen die folgenden Kriterien berücksichtigt werden:
schwere systemische Infektion
+
kein eindeutiger Erregernachweis, der das klinische Bild bereits erklärt
+
konventionelle Diagnostik eingeleitet
Zunächst ist eine Rücksprache mit dem mikrobiologischen Dienst (38094) erforderlich. Anschließend wird ein Auftrag über LAURIS erstellt. LAURIS-Aufträge ohne telefonische Absprache können nicht bearbeitet werden. Um eine zeitnahe Sequenzierung zu gewährleisten, ist eine frühzeitige Anmeldung (bis ca. 09:00) notwendig.
Die Blutabnahme muss mit einem speziellen Röhrchen erfolgen, das aktuell mit den auf Station verfügbaren Sarstedt-Blutabnahmesets nicht kompatibel ist. Nach Anmeldung in der Mikrobiologie erhalten Sie die erforderlichen Materialien per Rohrpost.
Nach ca. 24-48h wird das Ergebnis durch die Abteilung Mikrobiologie mitgeteilt.
Wir bitten um engmaschige Abstimmung mit dem ärztl. Dienst der Med. Mikrobiologie, damit der Zusatznutzen des Verfahrens bezüglich einer Verstetigung des Angebots evaluiert werden kann.
Für Rückfragen bezüglich Indikation/Interpretation wenden Sie sich bitte an die OÄ Dr. S. Zimmermann (38489) oder Dr. S. Klein (37975).

November 2024
Einführung Aspergillus-PCR
Zum 13.11.2024 erfolgt die Einführung einer PCR zum qualitativen Nachweis von Aspergillus-DNA. Diese ist für folgende Materialien validiert:
- BAL
- EDTA-Plasma
Zusätzlich kann in begründeten Einzelfällen eine Analyse von Bronchialsekret, Trachealsekret, Gewebeproben, Biopsien und Punktaten erfolgen, wenn eine Aspergillus-Infektion klinisch wahrscheinlich erscheint und mit anderen diagnostischen Verfahren nicht nachgewiesen werden konnte, der Assay ist hierfür jedoch nicht validiert. Abstriche oder Sputum sind nicht geeignet. Dieser PCR-Assay kann nur Aspergillus terreus auf Speziesebene differenzieren, eine Differenzierung der anderen Aspergillus-Spezies (z.B. A. fumigatus, A. flavus, A. niger) ist nicht möglich, die Ergebnisausgabe erfolgt nur auf Gattungs-Ebene (Aspergillus sp.). Der Test kann keine anderen Erreger (z.B. Mucorales, Fusarium, Scedosporium) von invasiven Schimmelpilz-Erkrankungen nachweisen. Eine molekulare Resistenztestung ist nicht möglich. Es ist daher immer in erster Linie ein kultureller Nachweis mit Resistenztestung anzustreben. Die Bestimmung des Aspergillus-Antigens (Galactomannan) unterstützt die Einschätzung der klinischen Wahrscheinlichkeit einer Infektion. Der neue PCR-Test dient als Ergänzung bei unklaren Situationen. Im Zweifel berät die/der diensthabende MikrobiologIn (38094).
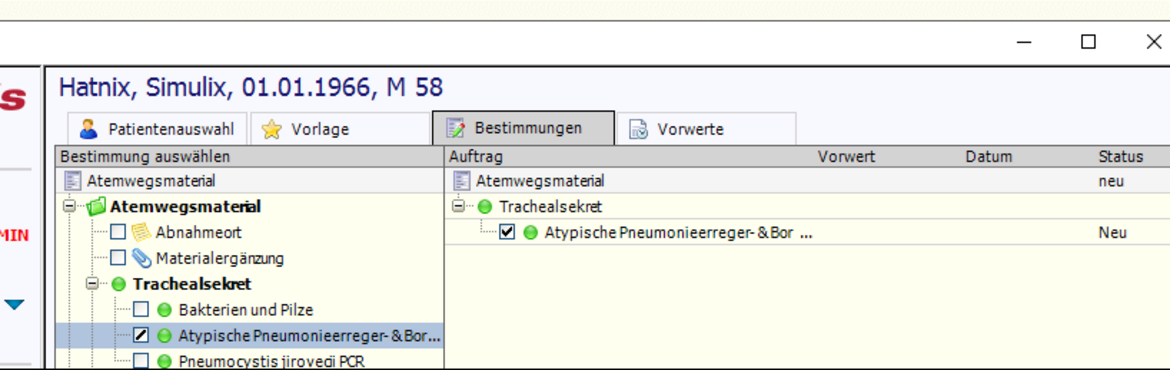
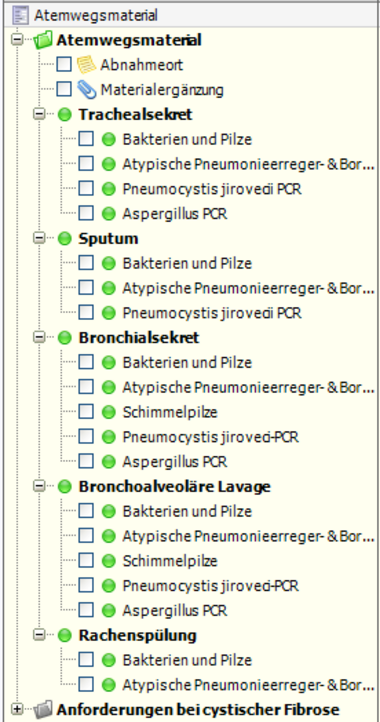
Die Anforderung erfolgt in LAURIS über Auftragserfassung > Zentrum für Infektiologie > Anforderung nach Material > Atemwegsmaterial > BAL (bzw. anderes Material) > Aspergillus PCR

April 2024
Umstellung von respiratorischen PCR-Assays für atypische Pneumonieerreger, Pertussis und Pneumocystis
Zum 8.4.2024 erfolgt eine Umstellung der respiratorischen PCR-Assays der medizinischen Mikrobiologie. Dies umfasst PCR-Panel für atypische Pneumonieerreger (Multiplex-PCR), Bordetella pertussis (Keuchhusten) und Pneumocystis jirovecii (PCP/PjP).
| bisheriges PCR-Panel atypische Pneumonieerreger | Atypische Pneumonieerreger- & Bordetella-PCR (Allplex™ PneumoBacter Assay) |
|
|
Die bisher als Einzelanforderung verfügbare PCR auf Bordetella pertussis/parapertussis wird in die Multiplex-PCR atypische Pneumonieerreger integriert, eine separate Anforderung ist nicht mehr möglich.
Zusätzlich wird durch die neue Multiplex-PCR auch Streptococcus pneumoniae und Haemophilus influenzae detektiert. Bei einer gezielten Diagnostik auf diese Erreger sollte jedoch weiterhin ein kultureller Nachweis zur Durchführung einer Resistenztestung angestrebt werden.
Weiterhin erfolgt ein Wechsel des Panels zum Nachweis und zur Quantifizierung von Pneumocystis jirovecii. Diese PCR wird weiterhin separat verfügbar sein. Kopienzahlen mit Resultaten vor dem 8.4. können nur bedingt mit den Resultaten ab dem 8.4. verglichen werden. Eine klare Tendenz zu niedrigeren oder höheren Kopienzahlen zwischen den beiden Assays konnte in einem Methodenvergleich jedoch nicht festgestellt werden.
Die Anforderung erfolgt in LAURIS über Auftragserfassung>Zentrum für Infektiologie>Anforderung nach Material>Atemwegsmaterial>Auswahl des spezifischen Materials>Atypische Pneumonieerreger- und Bordetella-PCR und/oder Pneumocystis jirovecii PCR.